本文介紹基於Tair Vector向量引擎實現化合物或藥物分子結構近似檢索的解決方案。
背景資訊
向量(Vector)檢索在AI製藥中扮演著至關重要的角色,在該方案中,通常以向量表示化合物和藥物,並通過向量空間中的相似性計算來預測、最佳化它們之間的相互作用。這種方案可以快速地篩選出具有優秀相互作用的化合物和藥物,從而加速新藥的研發進程。同時,向量檢索也能夠提高藥物篩選的準確度和效率,為醫藥研究人員提供更加高效、精確的藥物研究方法。
相比較傳統的向量檢索服務,Tair Vector的所有資料均在記憶體中,支援即時更新索引,具有更短的讀寫時延。同時,Vector提供的近鄰查詢(TVS.KNNSEARCH等)支援從資料庫中高效地擷取與目標分子結構最為相似的Top k個分子結構(k支援自訂),可降低人工誤操作或漏操作對專案造成不可控的風險。
方案概述
整體流程圖如下。
下載化學分子資料集。
本樣本使用的測試資料為PubChem開來源資料集,共計11012行資料,上述下載地址提供的資料為SMI格式,資料樣本如下,兩列分別為化學分子式和唯一ID。
說明在實際專案中,您可以寫入更多資料,體驗Tair毫秒層級檢索的特性。
CCC1=CN=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1ccc(OC)cc1OC,168000001 CC(C)CN1C(=O)C2SCCC2N2C(=S)NNC12,168000002 CC1=C[NH+]=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1cccc(C(F)(F)F)c1,168000003 CC1=CN=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1cccc(C(F)(F)F)c1,168000004若您從官方下載,則資料為SDF格式,您還需通過下述代碼將其轉換為SMI格式:
串連Tair執行個體,具體實現可參見範例程式碼中的
get_tair函數。在Tair中建立用於儲存分子結構的向量索引,具體實現可參見範例程式碼中的
create_index函數。寫入分子結構樣本資料,具體實現可參見範例程式碼中的
do_load函數。將通過RDKit庫提取分子結構資料的向量特徵,並通過Vector的TVS.HSET命令,將其唯一ID、特徵資訊和化學分子式存入Tair中。
進行相似分子結構查詢,具體實現可參見範例程式碼中的
do_search函數。將通過RDKit庫提取待查詢分子結構的向量特徵,然後通過Vector的TVS.KNNSEARCH命令,在Tair的指定索引中,查詢與該目標最為相似的分子結構。
樣本代碼
本樣本的Python版本為3.8,需提前安裝如下依賴庫:pip install numpy rdkit tair matplotlib。
import os
import sys
from tair import Tair
from tair.tairvector import DistanceMetric
from rdkit.Chem import Draw, AllChem
from rdkit import DataStructs, Chem
from rdkit import RDLogger
from concurrent.futures import ThreadPoolExecutor
RDLogger.DisableLog('rdApp.*')
def get_tair() -> Tair:
"""
串連Tair執行個體。
* host:Tair執行個體串連地址。
* port:Tair執行個體的連接埠號碼,預設為6379。
* password:Tair執行個體的密碼(預設帳號)。若通過自訂帳號串連,則密碼格式為“username:password”。
"""
tair: Tair = Tair(
host="r-bp1mlxv3xzv6kf****pd.redis.rds.aliyuncs.com",
port=6379,
db=0,
password="Da******3",
)
return tair
def create_index():
"""
建立用於儲存分子結構的向量索引:
* 本樣本的索引名稱為"MOLSEARCH_TEST"。
* 向量維度為512。
* 計算向量距離函數為L2。
* 索引演算法為HNSW。
"""
ret = tair.tvs_get_index(INDEX_NAME)
if ret is None:
tair.tvs_create_index(INDEX_NAME, 512, distance_type=DistanceMetric.L2, index_type="HNSW")
print("create index done")
def do_load(file_path):
"""
您需要輸入分子結構資料集的路徑,該方法會自動提取分子結構的向量特徵(smiles_to_vector),並將資料寫入Tair Vector中。
同時,該方法會調用parallel_submit_lines、handle_line、smiles_to_vector、insert_data等函數。
存入Tair的格式為:
* 向量索引名稱為“MOLSEARCH_TEST”。
* Key為分子結構的唯一ID,例如“168000001”。
* 特徵資訊為512維向量資訊。
* “smiles”為化學分子式,例如“CCC1=CN=C2C(C(=O)N(C)C(=O)N2C)/C1=N/c1ccc(OC)cc1OC”。
"""
num = 0
lines = []
with open(file_path, 'r') as f:
for line in f:
if line.find("smiles") >= 0:
continue
lines.append(line)
if len(lines) >= 10:
parallel_submit_lines(lines)
num += len(lines)
lines.clear()
if num % 10000 == 0:
print("load num", num)
if len(lines) > 0:
parallel_submit_lines(lines)
print("load done")
def parallel_submit_lines(lines):
"""
並發寫入的調度方法。
"""
with ThreadPoolExecutor(len(lines)) as t:
for line in lines:
t.submit(handle_line, line=line)
def handle_line(line):
"""
單個分子結構的寫入處理。
"""
if line.find("smiles") >= 0:
return
parts = line.strip().split(',')
try:
ids = parts[1]
smiles = parts[0]
vec = smiles_to_vector(smiles)
insert_data(ids, smiles, vec)
except Exception as result:
print(result)
def smiles_to_vector(smiles):
"""
提取分子結構的向量特徵,從SMI格式轉換為Vector。
"""
mols = Chem.MolFromSmiles(smiles)
fp = AllChem.GetMorganFingerprintAsBitVect(mols, 2, 512 * 8)
hex_fp = DataStructs.BitVectToFPSText(fp)
vec = list(bytearray.fromhex(hex_fp))
return vec
def insert_data(id, smiles, vector):
"""
將分子結構的Vector寫入Tair Vector中。
"""
attr = {'smiles': smiles}
tair.tvs_hset(INDEX_NAME, id, vector, **attr)
def do_search(search_smiles,k):
"""
您需要輸入待查詢分子結構,該方法會在Tair的指定索引中,查詢並返回與該目標最為相似的k個分子結構。
先提取待查詢分子結構的向量特徵,再通過TVS.KNNSEARCH命令查詢到k個(本樣本為10)最近的分子結構唯一ID,然後通過TVS.HMGET命令查詢到對應的分子式。
"""
vector = smiles_to_vector(search_smiles)
result = tair.tvs_knnsearch(INDEX_NAME, k, vector)
print("與查詢目標分子結構最為相似的10個分子結構如下:")
for key, value in result:
similar_smiles = tair.tvs_hmget(INDEX_NAME, key, "smiles")
print(key, value, similar_smiles)
if __name__ == "__main__":
# 串連Tair資料庫,並建立分子結構的向量索引,命名為“MOLSEARCH_TEST”。
tair = get_tair()
INDEX_NAME = "MOLSEARCH_TEST"
create_index()
# 寫入樣本資料。
do_load("D:\Test\Compound_168000001_168500000.smi")
# 在MOLSEARCH_TEST索引中,查詢與"CCOC(=O)N1CCC(NC(=O)CN2CCN(c3cc(C)cc(C)c3)C(=O)C2=O)CC1"最為相似的10個分子結構。
do_search("CCOC(=O)N1CCC(NC(=O)CN2CCN(c3cc(C)cc(C)c3)C(=O)C2=O)CC1",10)本樣本的正確執行結果如下:
create index done
load num 10000
load done
與查詢目標分子結構最為相似的10個分子結構如下:
b'168000009' 0.0 ['CCOC(=O)N1CCC(NC(=O)CN2CCN(c3cc(C)cc(C)c3)C(=O)C2=O)CC1']
b'168003114' 29534.0 ['Cc1cc(C)cc(N2CCN(CC(=O)NC3CCCC3)C(=O)C2=O)c1']
b'168000210' 60222.0 ['COc1ccc(N2CCN(CC(=O)Nc3cc(C)cc(C)c3)C(=O)C2=O)cc1OC']
b'168001000' 61123.0 ['COc1ccc(N2CCN(CC(=O)Nc3ccc(C)cc3)C(=O)C2=O)cc1OC']
b'168003038' 64524.0 ['CCN1CCN(c2cc(C)cc(C)c2)C(=O)C1=O']
b'168003095' 67591.0 ['O=C(CN1CCN(c2cccc(Cl)c2)C(=O)C1=O)NC1CCCC1']
b'168000396' 70376.0 ['COc1ccc(N2CCN(Cc3ccc(C)cc3)C(=O)C2=O)cc1OC']
b'168002227' 71121.0 ['CCOC(=O)CN1CCN(C2CC2)C(=O)C1=O']
b'168000441' 73197.0 ['Cc1cc(C)cc(NC(=O)CN2CCN(c3ccc(F)c(F)c3)C(=O)C2=O)c1']
b'168000561' 73269.0 ['Cc1cc(C)cc(N2CCN(CC(=O)Nc3ccc(C)cc3C)C(=O)C2=O)c1']結果展示
您也可以將相似分子結構繪製成圖片,樣本如下。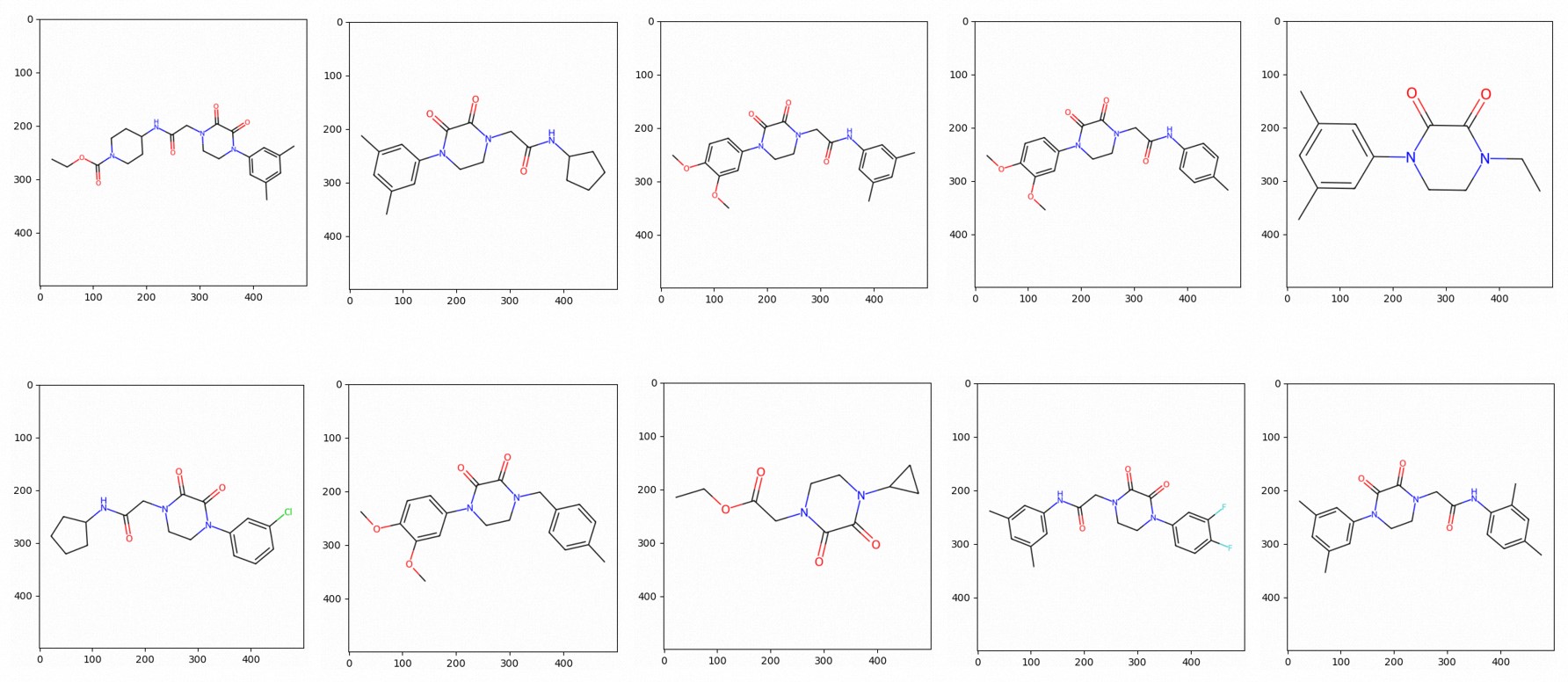
總結
使用Tair Vector檢索待分析的分子結構,可以在毫秒層級檢索到最相似的分子結構列表。且隨著時間累積,Tair資料庫中會儲存越來越多的分子結構資料集,這使得後續的查詢請求更加準確與及時。該方案將降低藥物研發領域的研發耗時,提升整體研發效率。