本文以AutoDock Vina軟體為例,介紹如何在E-HPC上進行高效能運算實現虛擬藥物篩選。
背景資訊
分子對接(Molecular docking)是虛擬藥物篩選中的關鍵環節。AutoDock Vina作為一款開源的分子對接軟體,具有速度快、演算法準確等優點,特別適用於搭建基於分子對接的虛擬篩選,它基於MGLTools工具包進行使用。MGLTools包括AutoDock Tools(ADT)和Python Molecular Viewer(PMV)。ADT用來為Vina產生輸入檔案,PMV用來查看結果。更多資訊,請參見AutoDock Vina和MGLTools。
本文通過類比小分子配體和生物大分子受體相互作用的過程,預測配體與受體的結合模式和親和力,類比實現對藥物的篩選。目前商業應用較廣泛的Specs、Enamine和ChemDiv化合物庫,均可提供大量配體類比計算配體和給定受體的相互作用。由於不同配體之間沒有依賴,因此可以大規模平行處理。本文同樣適用於其它大批量、高並發處理需求的生物、醫藥等情境。
準備工作
建立E-HPC叢集。
具體操作,請參見使用嚮導建立叢集。您需要注意以下配置參數:
參數
說明
硬體參數
部署方式為標準,包含2個管控節點,1個計算節點和1個登入節點。
節點均採用ecs.c7.large執行個體規格,配置為2 vCPU,4 GiB記憶體,Ice Lake處理器,2.7 GHz。
軟體配置
鏡像選擇CentOS 7.6公用鏡像,調度器選擇pbs,開啟VNC。
建立叢集使用者。具體操作,請參見建立使用者。
叢集使用者用於登入叢集,進行編譯軟體、提交作業等操作。本文建立的使用者樣本如下:
使用者名稱:vinatest
使用者組:sudo許可權組
安裝軟體。具體操作,請參見安裝軟體。
需安裝的軟體如下:
vina,版本為1.1.2。
AutoDockTools,版本為1.5.7。
intel-mpi,版本為2018。
步驟一:串連叢集
選擇以下一種方式串連叢集。本文使用的使用者名稱為vinatest,串連叢集後會預設登入到/home/vinatest。
通過用戶端
該方式僅支援使用PBS調度器的叢集。操作前,請確保您已下載安裝E-HPC用戶端,且已配置用戶端所需環境。具體操作,請參見配置用戶端所需環境。
開啟並登入E-HPC用戶端。
在用戶端左側導覽列,單擊會話管理。
在會話管理頁面的右上方,單擊terminal,開啟Terminal視窗。
通過控制台
登入彈性高效能運算控制台。
在頂部功能表列左上方處,選擇地區。
在左側導覽列,單擊叢集。
在叢集頁面,找到目的地組群,單擊遠端連線。
在遠端連線頁面,輸入集群使用者名稱、登入密碼和連接埠,單擊ssh串連。
步驟二:提交作業
下載並解壓作業檔案。
下載作業檔案。
說明如果叢集中沒有安裝git,請先執行
sudo yum install -y git安裝git。git clone https://code.aliyun.com/best-practice/022.git解壓作業檔案。
cd 022 tar xzvf vina-ehpcarrayjob.tar.gz
修改設定檔。
cd vina-ehpcarrayjob vim conf.txt在conf.txt中,刪除以下兩行內容:
receptor = 1fkn_rgd.pdbqt ligand = $file建立作業指令碼,命名為vina.sh。
cd /home/vinatest vim vina.sh指令碼內容如下:
#!/bin/bash #PBS -N vina_job #PBS -l nodes=1:ppn=2 #PBS -l walltime=00:10:00 #PBS -j oe #PBS -v receptor="022/vina-ehpcarrayjob/1fkn_rgd.pdbqt",ligand="022/vina-ehpcarrayjob/test/ligand_1.pdbqt",config="022/vina-ehpcarrayjob/conf.txt" cd $PBS_O_WORKDIR ppn="$NCPUS" cd $PBS_O_WORKDIR vina --receptor $receptor --config $config --ligand $ligand --out out.pdbqt --cpu $ppn提交作業。
qsub vina.sh預期返回如下,表示產生的作業ID為0.scheduler。
0.scheduler
步驟三:查看結果
查看作業運行情況。
qstat -x 0.scheduler預期返回如下,當返回資訊中
S為R時,表示作業正在運行中;當返回資訊中S為F時,表示作業已經運行結束。Job id Name User Time Use S Queue ---------------- ---------------- ---------------- -------- - ----- 0.scheduler vina_job vinatest 00:01:56 F workq說明作業運行結束後會產生結果檔案out.pdbqt,儲存在提交作業時的路徑下,本文樣本為
/home/vinatest/out.pdbqt。使用VNC可視化查看作業結果。
開啟VNC。
說明請確保叢集所屬安全性群組已開啟VNC所需連接埠。控制台操作時系統會自動開啟12016連接埠,用戶端操作時,請您自行開啟連接埠,首個VNC視窗使用12017連接埠,如果有多個使用者使用VNC,則連接埠號碼按順序遞增。
通過用戶端
在用戶端的左側導覽列,單擊會話管理。
在會話管理頁面的右上方,單擊VNC,開啟VNC視窗。
通過控制台
在彈性高效能運算控制台的左側導覽列,單擊叢集。
在叢集頁面,找到目的地組群,單擊更多 > VNC。
使用VNC遠端連線可視化服務。具體操作,請參見串連可視化服務。
在VNC視窗中,選擇Application>System Tools>Terminal。
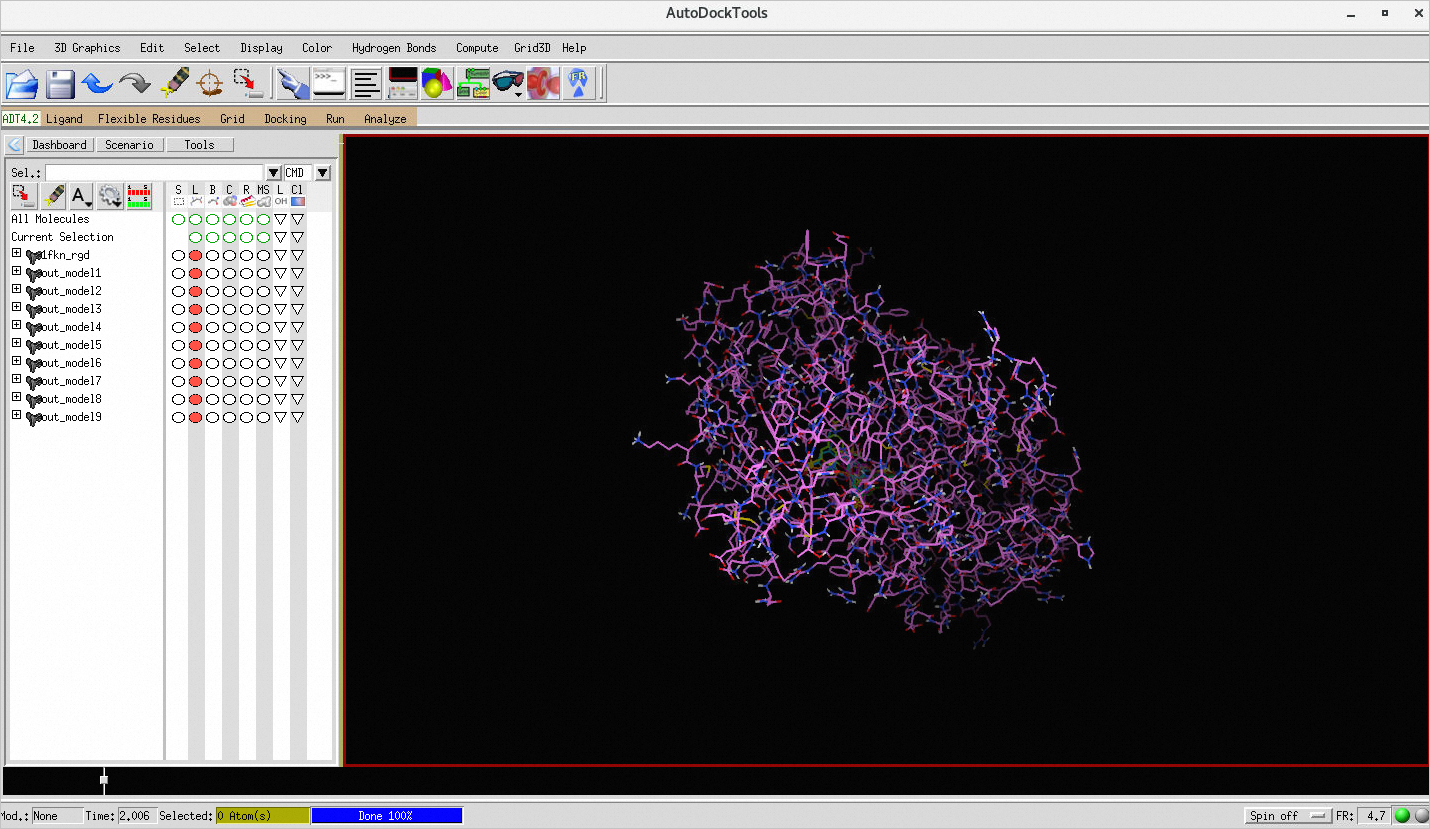
在Terminnal中執行以下命令,開啟AutoDock Tools並載入作業結果。
/usr/local/bin/adt /home/vinatest/022/vina-ehpcarrayjob/1fkn_rgd.pdbqt /home/vinatest/out.pdbqt在彈出的AutoDock Tools視窗中載入模型後,顯示結果如下: