在病原体鉴定过程中,通过对比待测样本与已知病原体基因组数据库,可以高效准确的对待测样本进行鉴定分析。AGS服务提供了针对宏基因组测序数据的快速比对能力。本文介绍如何通过AGS排查待测病原体。
前提条件
已提交阿里云基因服务公测申请,加入公测白名单。
如果您使用的是RAM用户,公测申请时请提供RAM用户的UID。请登录账号管理控制台查询UID。
背景信息
新冠肺炎发病后数周内都可以在上呼吸道或下呼吸道中检测到新型冠状病毒SARS-CoV-2 RNA(以下简称“新冠病毒”)。目前,已经有众多新冠病毒RT-PCR试剂盒可供选择,虽然PCR操作成本低、实效性高。但由于受病毒浓度和试剂盒质量等因素的影响,容易出现假阴性,需要多次检测确认,而且一次检测只能对一种特定病毒进行排查。
泛病原体检测的基因组下一代测序(Metagenomics Next-generation Sequencing, mNGS)技术直接从临床样本中随机抽取一定比例的核酸片段(包括大量人源核酸和少量微生物核酸)进行测序、数据库比对和生物信息分析,进而对病原微生物进行无偏性鉴定。该技术为新冠病毒SARS-CoV-2 RNA的早期发现和准确测序做出很大贡献。
相较于RT-PCR试剂盒检测,虽然mNGS方式检测分析周期相对较长,但准确率高,检测全面,可以一次排查多种病毒,同时也可监测病毒在传播过程中可能发生的变异,增强对病毒的防控。临床上,针对荧光定量PCR无法确诊的疑似患者进行二次检测可进一步提升检测结果的准确性,有效防止病毒变异产生的漏检。基于核酸序列比对的分析方式,一旦病原体的基因组已知,通过更新数据库,就可以实现高效准确地检测病原体。
阿里云基因计算服务AGS提供了针对mNGS宏基因组测序数据的快速比对能力,对一组肺泡采样测序的宏基因组数据3.2Gbase(22M reads),60秒内可以完成和已知的病原体基因组包括新型冠状病毒SARS-CoV-2,39种BetaCov RNA,以及9334种已知病毒的参考序列的比对,并且支持自定义的病毒库的上传和比对。对于疾控中心,医院,实验室只需要一个阿里云的对象存储Bucket, 以及命令行ags就可以完成整个的比对过程,并拿到高质量的匹配reads的数据和初步质量报告,为多种病原体检测,进一步的新冠病毒的蛋白质研究和变异研究提供了快捷准确的数据支撑。
欢迎基因测序厂商,疾控中心,医院,学校,制药企业申请使用。
准备工作
配置AGS。AGS的下载和安装,请参见AGS命令行帮助。
命令示例:
ags config init下载安装ossutil命令行,详情请参见安装ossutil。
注册阿里云账号,开通对象存储OSS服务并创建存储空间(Bucket)用于存放mNGS测序数据(例如:oss://my-test-shenzhen)。开通OSS请参见开通OSS服务。
说明如果您使用的是RAM用户,建议您重新创建OSS Bucket,以确保该账号是所使用Bucket的Owner。执行以下命令确认Bucket的Owner。
ossutil stat oss://<your new bucket name>为服务AGS服务授予Bucket的GetBucketInfo权限。
说明请确保指定Bucket的Owner是您当前的账号,否则建议您新建一个Bucket后再进行GetBucketInfo的授权。
如果您使用的是RAM用户,需要为RAM用户授予AliyunOSSFullAccess权限,请参见为RAM用户授权。
命令示例:
ags config oss <bucket_name>
rna-mapping使用示例
以下操作将为您演示如何通过命令检测AGS rna-mapping病毒。
与新冠病毒的比对
运行ossutil命令,将mNGS测序数据上传至存储空间(Bucket)。
命令示例:
ossutil cp ICU6G_S2_L001_R1_001.fastq.gz oss://my-test-shenzhen/cov2-samples/ ossutil cp ICU6G_S2_L001_R2_001.fastq.gz oss://my-test-shenzhen/cov2-samples/运行比对任务,对比mNGS数据和已知RNA序列数据。
本文以比对ICU6G_S2_L001测序样本和新冠病毒的相似度为例,运行任务如下。
命令格式:
Usage: ags remote run rna-mapping \ # <rna-mapping>: RNA 序列的比对任务 --region <region_id> \ # <cn-shenzhen|cn-beijing|...>: 地域ID,目前支持深圳和北京。 --bucket <bucket_name> \ # <bucket_name> 对象存储bucket的名称 --fastq1 <path_fq1> \ # 双端测序数据fq1相对路径 --fastq2 <path_fq2>\ # 双端测序数据fq2的相对路径 --output-bam <path_of_output_bam> \ #产出比对结果bam的输出路径,报告也在同样位置,以.txt结尾 --reference [sars-cov-2 | betacov-ncbi-39 | viral-9334 | <path of RNA library reference in specified bucket >] # 参考序列预置了新型冠状病毒sars-cov-2和目前已经知道的39种betacov的冠状病毒,可以指定自定义的病毒序列库命令示例:
ags remote run rna-mapping \ --region cn-shenzhen \ --fastq1 cov2-samples/ICU6G_S2_L001_R1_001.fastq.gz \ --fastq2 cov2-samples/ICU6G_S2_L001_R2_001.fastq.gz \ --bucket my-test-shenzhen \ --output-bam bam/ICU6G_S2.bam \ --reference sars-cov-2 INFO[0002] {"JobName":"rna-mapping-gpu-2ms6w"} INFO[0002] Job submit succeed检查比对任务和比对结果。
在这个比对任务示例中,10M reads和新冠病毒序列MN908947.3比对产生了3629个高质量重合的reads,并在新冠病毒特征区间排列分(AS)超过120分的序列(reads)条数有404个。说明可以精确的从此样本的测序数据中检测出SARS-CoV-2 RNA的序列。
比对结果示例:
ags remote get rna-mapping-gpu-2ms6w --show +-----------------------+------------------+-----------+-------------------------------+----------+-------------------------------+-------------+-------------+ | JOB NAME | JOB NAMESPACE | STATUS | CREATE TIME | DURATION | FINISH TIME | TOTAL READS | TOTAL BASES | +-----------------------+------------------+-----------+-------------------------------+----------+-------------------------------+-------------+-------------+ | rna-mapping-gpu-2ms6w | XXXXXXXXXXXX | Succeeded | 2020-03-04 16:40:30 +0800 CST | 43s | 2020-03-04 16:41:13 +0800 CST | 10369818 | 1456539874 | +-----------------------+------------------+-----------+-------------------------------+----------+-------------------------------+-------------+-------------+ +---------------------------------+--------------------------------------------+ | JOB DETAIL | | +---------------------------------+--------------------------------------------+ | rna_matached_reads | 480 | | rna_is_sars_cov2 | True | | rna_mapping_oss_region | cn-shenzhen | | rna_mapping_fastq_second_name | cov2-samples/ICU6G_S2_L001_R2_001.fastq.gz | | rna_mapping_no_unmapped | | | rna_mapping_service | s | | rna_matached_reads_alignment | 404 | | rna_high_quality_mapped | 3629 | | rna_mapping_fastq_first_name | cov2-samples/ICU6G_S2_L001_R1_001.fastq.gz | | rna_mapping_mark_dup | | | rna_mapping_reference_file_name | sars-cov-2 | | rna_cov_detail_file | bam/ICU6G_S2.bam.cov.txt | | rna_mapping_bam_file_name | bam/ICU6G_S2.bam | | rna_mapping_bucket_name | my-test-shenzhen | +---------------------------------+--------------------------------------------+执行ossutil命令,下载对比数据和简单报告。
命令示例:
ossutil ls oss://my-test-shenzhen/bam/ICU6G_S2.bam LastModifiedTime Size(B) StorageClass ETAG ObjectName 2020-03-04 16:41:11 +0800 CST 356320 Standard 9596D012A30438A0073A2A0B38F5D578 oss://my-test-shenzhen/bam/ICU6G_S2.bam 2020-03-04 16:41:11 +0800 CST 2889 Standard 63175E7180D110BA9D3BAB34F4313C59 oss://my-test-shenzhen/bam/ICU6G_S2.bam.cov.txt 2020-03-04 16:41:11 +0800 CST 396 Standard 940D51FF7ECFF60B5E5A41D1F635180D oss://my-test-shenzhen/bam/ICU6G_S2.bam.summary.json ossutil cp oss://my-test-shenzhen/bam/HKU2_160660.summary.json . ossutil cp -r oss://my-test-shenzhen/bam/ICU6G_S2.bam.cov.txt . ossutil cp oss://my-test-shenzhen/bam/HKU2_160660.bam .报告示例:
cat bam/ICU6G_S2.bam.cov.txt Summary: High Quality Mapped Reads is: 3629 Matched reads in orf1ab range is: 480 Matched reads in orf1ab range with alignment score (AS) is greater than 120: 404 /data/cov2-samples_ICU6G_S2_L001_R1_001.fastq.gz-output/ICU6G_S2.bam is similar to SARS-CoV-2 with very high mappQ and AS reads: True 21571 21581 21591 21601 21611 21621 21631 NNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN ATGTTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC ATGTT GTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC ATGTTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGT CAATTACCCCCTGC ATGTTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGA CCCCCTGC ATGTTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC ATGTTTGTTTTTCTTGTTTTATTGCCA agtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc atgtttgtttttcttgttttattgcca AGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC ATGTTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc ATGTTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC ATGTTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc ATGTTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc ATGTTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC ATGTTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC atgtttgtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc TGTTTGTTTTTCTTGTTTT CACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC tgtttgtttttcttgtttt TTTGTTTTTCTTGTTTTATTGCCACTAGTCTCTAGTCAGTGTGTTAATCTTACAACCAGAACTCAATTACCCCCTGC gtttttcttgttttattgccactagtctctagtcagtgtgttaatcttacaaccagaactcaattaccccctgc进一步分析比对数据。
您可以通过samtools stats, plot-bamstat 等工具将比对数据进一步分析coverage, depth等的相似度,还可以进一步实现蛋白质组成分析,以及变异分析。
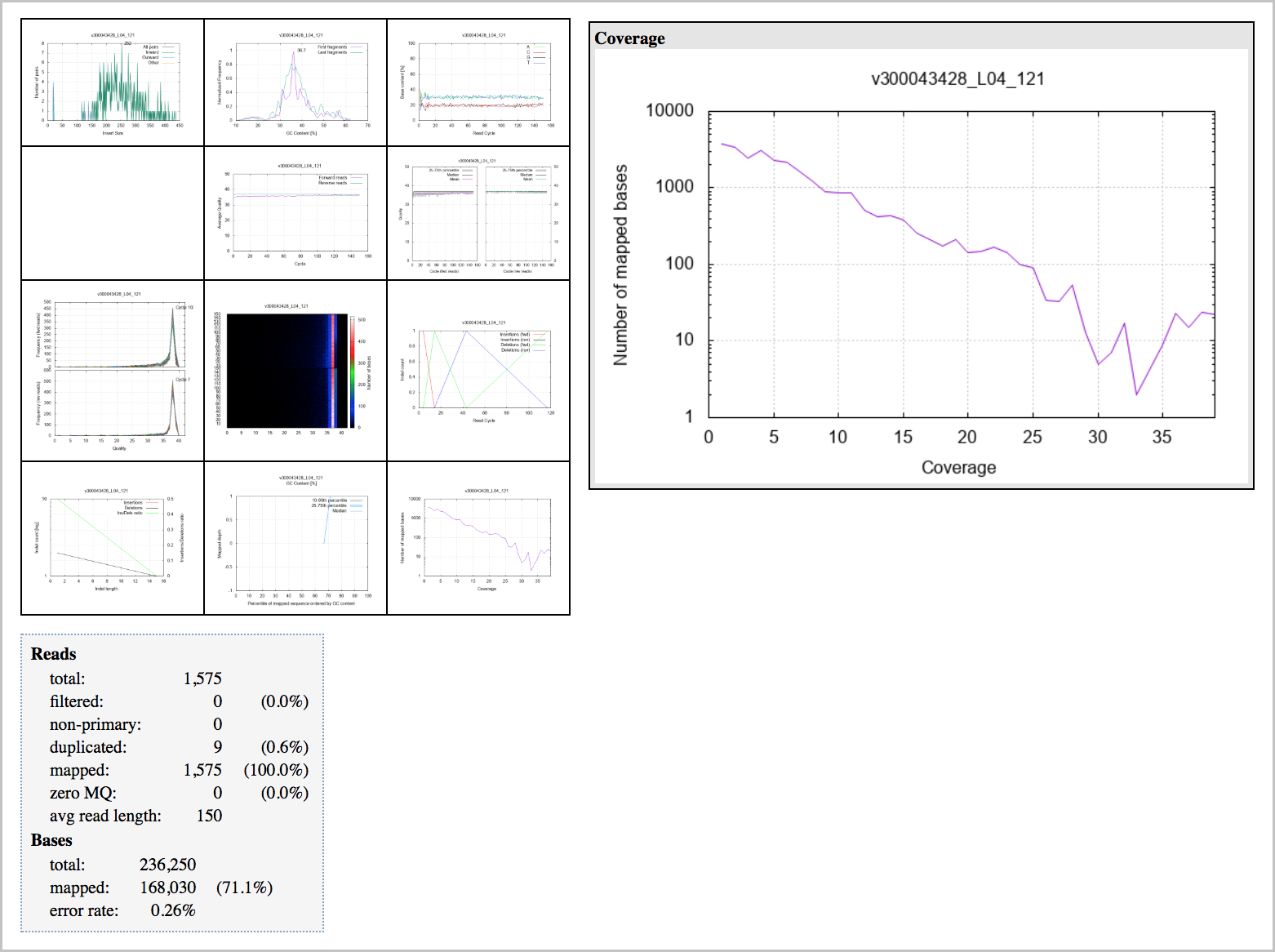

重复上述步骤可实现不同样本之间的比对。
与已知的39个betaCov病毒的比对
运行ossutil命令,将mNGS测序数据上传至存储空间(Bucket)。
命令示例:
ossutil cp ICU6G_S2_L001_R1_001.fastq.gz oss://my-test-shenzhen/cov2-samples/ ossutil cp ICU6G_S2_L001_R2_001.fastq.gz oss://my-test-shenzhen/cov2-samples/运行对比任务。
命令示例:
ags remote run rna-mapping \ --region cn-shenzhen \ --fastq1 cov2-samples/ICU6G_S2_L001_R1_001.fastq.gz \ --fastq2 cov2-samples/ICU6G_S2_L001_R2_001.fastq.gz \ --bucket my-test-shenzhen \ --output-bam bam/ICU6G_S2_virus.bam \ --reference betacov-ncbi-39 INFO[0011] {"JobName":"rna-mapping-gpu-6mpcc"} INFO[0011] Job submit succeed检查比对任务和比对结果。
比对结果示例:
ags remote get rna-mapping-gpu-6mpcc --show +-----------------------+------------------+-----------+-------------------------------+----------+-------------------------------+-------------+-------------+ | JOB NAME | JOB NAMESPACE | STATUS | CREATE TIME | DURATION | FINISH TIME | TOTAL READS | TOTAL BASES | +-----------------------+------------------+-----------+-------------------------------+----------+-------------------------------+-------------+-------------+ | rna-mapping-gpu-6mpcc | XXXXXXXXX | Succeeded | 2020-03-04 17:36:21 +0800 CST | 40s | 2020-03-04 17:37:01 +0800 CST | 10369818 | 1456539874 | +-----------------------+------------------+-----------+-------------------------------+----------+-------------------------------+-------------+-------------+ # 2014 mapped reads detected, but no mapped reads found in range +---------------------------------+--------------------------------------------+ | JOB DETAIL | | +---------------------------------+--------------------------------------------+ | rna_mapping_reference_file_name | betacov-ncbi-39 | | rna_matached_reads_alignment | 0 | | rna_mapping_bam_file_name | bam/ICU6G_S2_virus.bam | | rna_mapping_fastq_first_name | cov2-samples/ICU6G_S2_L001_R1_001.fastq.gz | | rna_mapping_oss_region | cn-shenzhen | | rna_cov_detail_file | bam/ICU6G_S2_virus.bam.cov.txt | | rna_mapping_no_unmapped | | | rna_matached_reads | 0 | | rna_mapping_mark_dup | | | rna_mapping_service | s | | rna_high_quality_mapped | 2014 | | rna_mapping_bucket_name | my-test-shenzhen | | rna_mapping_fastq_second_name | cov2-samples/ICU6G_S2_L001_R2_001.fastq.gz | | rna_is_sars_cov2 | False | +---------------------------------+--------------------------------------------+
与自定义病毒库的比对
从NCBI GeneBank下载reference序列合并成为一个多contig的参考序列。
示例:
搜索核酸包含“betacov”的所有参考系列, 并下载参考系列。
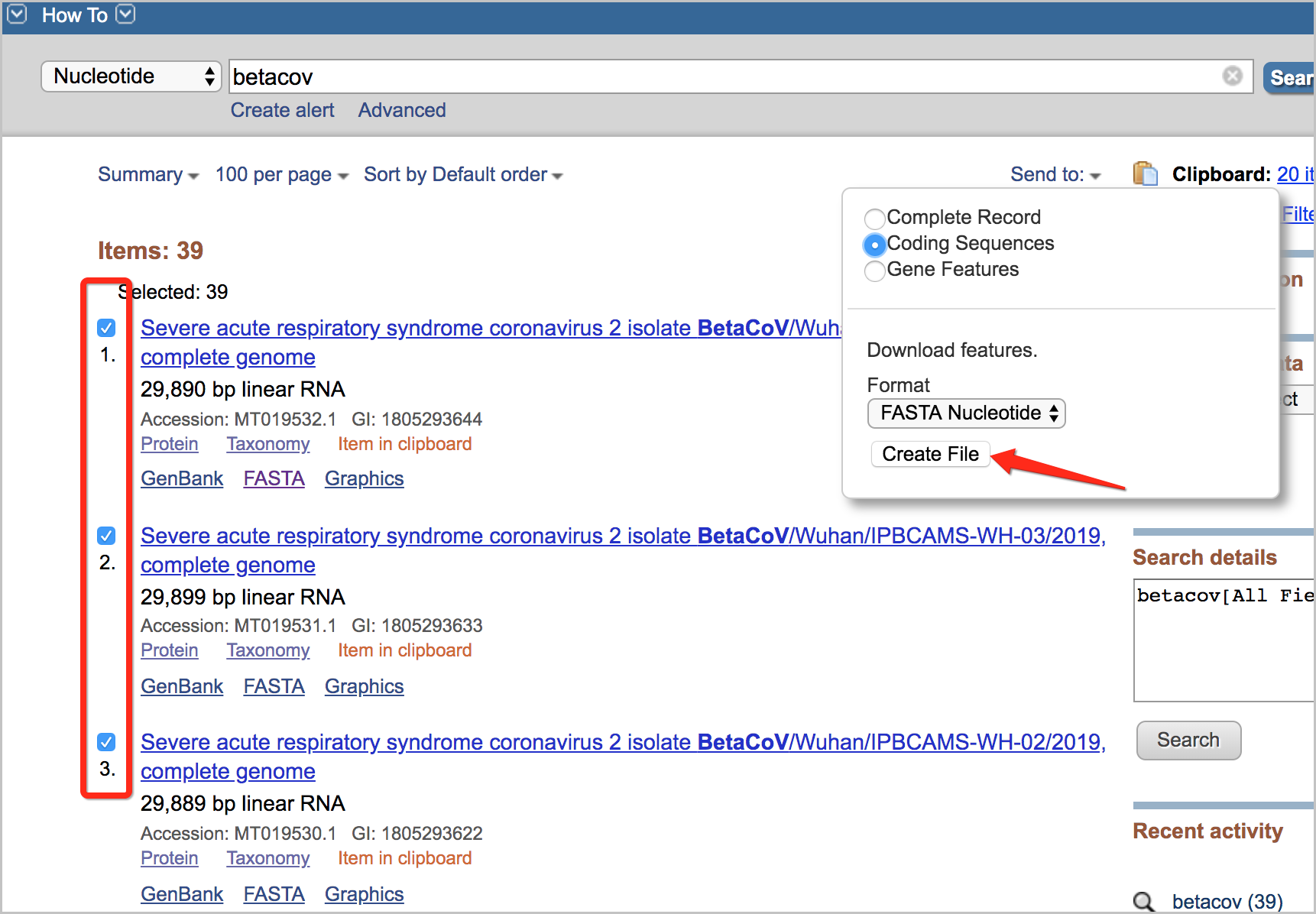
把下载的序列文件
sequence.fa改名为betacov-ncbi-test.fa。上传reference到存储空间。
命令示例:
ossutil cp betacov-ncbi-test.fa oss://my-test-shenzhen/ref/提交比对任务,指定reference的路径。
命令示例:
ags remote run rna-mapping \ --region cn-shenzhen \ --fastq1 cov2-samples/ICU6G_S2_L001_R1_001.fastq.gz \ --fastq2 cov2-samples/ICU6G_S2_L001_R2_001.fastq.gz \ --bucket my-test-shenzhen \ --output-bam bam/ICU6G_S2_virus.bam \ --reference ref/betacov-ncbi-test.fa INFO[0002] {"JobName":"rna-mapping-gpu-69mwb"} INFO[0002] Job submit succeed查看比对报告和获取匹配的比对数据。
对比结果示例:
ags remote get rna-mapping-gpu-69mwb --show +-----------------------+------------------+-----------+-------------------------------+----------+-------------------------------+-------------+-------------+ | JOB NAME | JOB NAMESPACE | STATUS | CREATE TIME | DURATION | FINISH TIME | TOTAL READS | TOTAL BASES | +-----------------------+------------------+-----------+-------------------------------+----------+-------------------------------+-------------+-------------+ | rna-mapping-gpu-69mwb | 1365606736606053 | Succeeded | 2020-03-04 17:47:00 +0800 CST | 40s | 2020-03-04 17:47:40 +0800 CST | 10369818 | 1456539874 | +-----------------------+------------------+-----------+-------------------------------+----------+-------------------------------+-------------+-------------+ +---------------------------------+--------------------------------------------+ | JOB DETAIL | | +---------------------------------+--------------------------------------------+ | rna_mapping_fastq_first_name | cov2-samples/ICU6G_S2_L001_R1_001.fastq.gz | | rna_mapping_fastq_second_name | cov2-samples/ICU6G_S2_L001_R2_001.fastq.gz | | rna_mapping_mark_dup | | | rna_mapping_oss_region | cn-shenzhen | | rna_cov_detail_file | bam/ICU6G_S2_virus.bam.cov.txt | | rna_is_sars_cov2 | False | | rna_mapping_bam_file_name | bam/ICU6G_S2_virus.bam | | rna_mapping_service | s | | rna_matached_reads_alignment | 0 | | rna_high_quality_mapped | 2014 | | rna_mapping_bucket_name | my-test-shenzhen | | rna_mapping_no_unmapped | | | rna_mapping_reference_file_name | ref/betacov-ncbi-test.fa | | rna_matached_reads | 0 | +---------------------------------+--------------------------------------------+ +---------------------------------+------------------------------------------+下载比对数据做进一步分析。
命令示例:
ossutil ls oss://my-test-shenzhen/bam/ICU6G_S2_virus.bam LastModifiedTime Size(B) StorageClass ETAG ObjectName 2020-03-04 17:47:38 +0800 CST 753458 Standard DF7B1A6CA5AF5DE6BF4FFDBB6DEF71C3 oss://my-test-shenzhen/bam/ICU6G_S2_virus.bam 2020-03-04 17:47:38 +0800 CST 1474 Standard 9D7968A779A0DE7C1993CC2A8D0E5A56 oss://my-test-shenzhen/bam/ICU6G_S2_virus.bam.cov.txt 2020-03-04 17:47:38 +0800 CST 397 Standard 81170E30BAAFEB947A2238E015171A51 oss://my-test-shenzhen/bam/ICU6G_S2_virus.bam.summary.json Object Number is: 3 ossutil cp oss://my-test-shenzhen/bam/ICU6G_S2_virus.bam.summary.json . cat bam/ICU6G_S2_virus.bam.summary.json { "total_reads":10369818, "total_bases":1456539874, "pass_vendor_filter_reads":10369818, "mapped_reads":6736, "pair_reads":6680, "properly_paired_reads":6520, "mapq_40_to_inf_reads":2030, "mapq_30_to_40_reads":0, "mapq_20_to_30_reads":1, "mapq_10_to_20_reads":3, "mapq_0_to_10_reads":23, "mapq_0_reads":10367761, "GC":"46.499%", "total_alignment":2057, "supplementary_alignment":0 }% ossutil cp oss://my-test-shenzhen/bam/ICU6G_S2_virus.bam . samtools view bam/ICU6G_S2_virus.bam
